python数据分析之用sklearn预测糖尿病
目录
- 一、数据集描述
- 二、准备工作
- 三、实验环境和工具
- 四、预测分析
- 4.1探索性数据分析
- 五、可视化分析
- 六、构建baseline
- 七、数据预处理
- 八、离群值处理
- 九、缺失值处理
- 十、特征工程
- 十一、数据标准化
- 十二、模型构建与调参优化
- 十三、总结
一、数据集描述
本数据集内含十个属性列
Pergnancies: 怀孕次数
Glucose:血糖浓度
BloodPressure:舒张压(毫米汞柱)
SkinThickness:肱三头肌皮肤褶皱厚度(毫米)
Insulin:两个小时血清胰岛素(μU/毫升)
BMI:身体质量指数,体重除以身高的平方
Diabets Pedigree Function: 疾病血统指数
是否和遗传相关,Height:身高(厘米)
Age:年龄
Outcome:0表示不患病,1表示患病。
任务:建立机器学习模型以准确预测数据集中的患者是否患有糖尿病
二、准备工作
查阅资料得知各属性的数据值要求,方面后期对于数据的分析与处理过程。
属性列名称 数据值要求
Pergnancies(怀孕次数) 符合常理即可(可为0)
Glucose(血糖浓度) 正常值为:80~120
BloodPressure(舒张压(毫米汞柱)) 正常值为:60~80
SkinThickness(肱三头肌皮肤褶皱厚度(毫米)) 不为0
Insulin(两个小时血清胰岛素(/毫升)) 正常值为:35~145
BMI(身体质量指数:体重除以身高的平方) 正常值为:18.5~24.9
Diabets Pedigree Function:(疾病血统指数:是否和遗传相关) 无特殊值要求
Height(身高(厘米)) 不为0 符合常理即可
Age(年龄) 符合常理即可
Outcome(0表示不患病,1表示患病) 标签值
三、实验环境和工具
python3.5.6 + jupyter
数据处理 pandas、numpy
可视化 matplotlib、seaborn
模型构建 sklearn
四、预测分析
4.1探索性数据分析
数据描述
首先观察基本的数据类型,以及数据是否存在缺失情况,简要统计信息
all_data.shape all_data.info()
<class 'pandas.core.frame.DataFrame'> RangeIndex: 768 entries, 0 to 767 Data columns (total 10 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 Pregnancies 768 non-null int64 1 Glucose 768 non-null int64 2 BloodPressure 768 non-null int64 3 SkinThickness 768 non-null int64 4 Insulin 768 non-null int64 5 BMI 768 non-null float64 6 DiabetesPedigreeFunction 768 non-null float64 7 Age 768 non-null int64 8 Height 766 non-null object 9 Outcome 768 non-null int64 dtypes: float64(2), int64(7), object(1) memory usage: 60.1+ KB
数据总量时比较少的只有768个例子,可以看到除Height外的属性都为数值型属性。在后续数据预处理过程需要对Height属性进行类型转换操作。目前没有缺失值的出现。
# height 数值类型 为object 需要转化为 数值型
all_data = all_data.astype({'Height':'float64'})
all_data.describe()
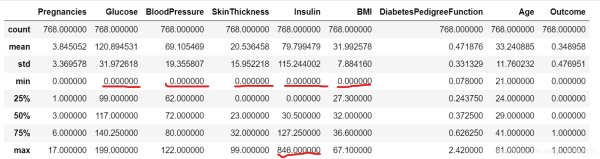
发现两个问题:
1.缺失值
从其中的min值可以很直观地观察到,Glucose, BloodPressure, SkinTinckness, Insulin, BMI等特征存在0值的情况(当然Pregnancies根据常识判断是可以为0的)。而根据常规范围明显可以判定这些0值是不合理的,所以也是一种缺失值缺失值,后续数据预处理需要对这些缺失值进行填充处理。
2.离群值/异常值
Glucose,BloodPressure,SkinTinckness,Insulin等特征的max值和75%分位点值或者min值和25%分位点值之间的差距比较大,初步判断可能存在离群值/异常值。尤其是SkinThickness和Insulin特征(具体见图4红色框部分),后续可以通过可视化进一步直观地观察判断。
为了方便后序对缺失值的统一处理,将异常值统一替换为np.nan。
import numpy as np
#缺失值替换 经分析,除怀孕次数,其他特征的0值表示缺失值 替换为np.nan
replace_list = ['Glucose', 'BloodPressure', 'SkinThickness', 'Insulin', 'BMI', 'Height']
all_data.loc[:,replace_list] = all_data.loc[:,replace_list].replace({0:np.nan})
#各特征缺失数量统计
null_count = all_data.isnull().sum().values
# 缺失值情况
plt.figure()
sns.barplot(x = null_count, y = all_data.columns)
for x, y in enumerate(null_count):
plt.text(y, x, "%s" %y, horizontalalignment='center', verticalalignment='center')
plt.show()
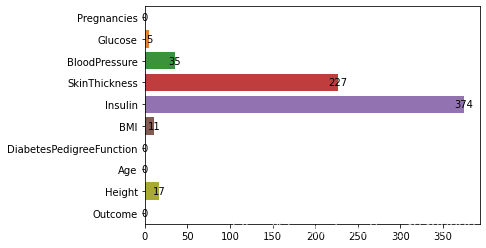
可以观察到Glucose,Insulin,SkinThickness,BMI,Height等特征都存在缺失值。并且 Insulin,SkinThickness缺失值比较多,分别占到了48%,30%的比例。所以后期数据预处理也是很关键的。
五、可视化分析
接下来通过更多针对性的可视化,来进一步探索特征值的分布以及特征和预测变量之间的关系
# 患病和不患病情况下 箱线图查看数据分散情况
for col in all_data.columns:
plt.figure(figsize = (10,6))
if all_data[col].unique().shape[0] > 2:
sns.boxplot(x="Outcome", y=col, data=all_data.dropna())
else:
sns.countplot(col,hue = 'Outcome',data = all_data.dropna())
plt.title(col)
plt.show()
部分输出:
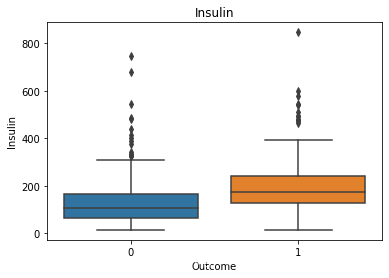
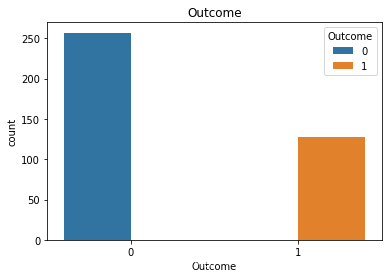
观察患病和不患病情况下 各特征值或者人数分布 label接近2:1 存在一定的分布不平衡 像insulin之类的特征离群值是比较多的,由于离群值会对模型评估产生影响,所以后续可能要做处理,剔除偏离较大的离群值
# 患病和不患病情况下 各特征的分布情况
for col in all_data.drop('Outcome',1).columns:
plt.figure()
sns.displot(data = all_data, x = col,hue = 'Outcome',kind='kde')
plt.show()
部分输出:
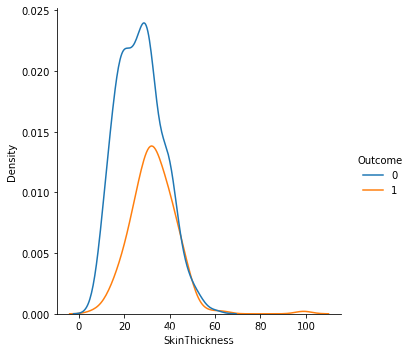
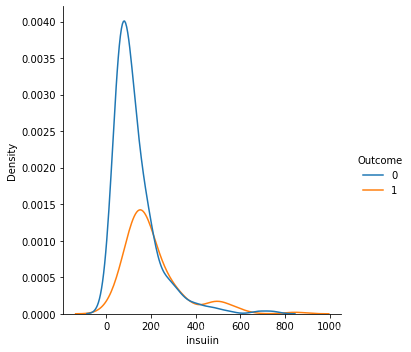
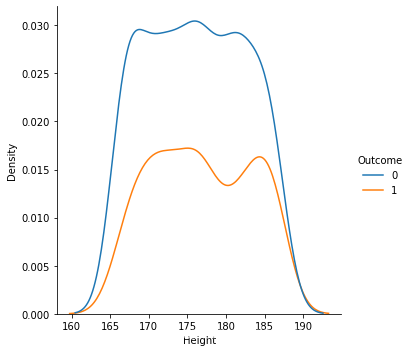
1.从数据样本本身出发研究数据分布特征,可以发现在患病和不患病两种情况下,部分特征的密度分布比较相近,特别是height的分布图,发现两曲线基本相近。感觉和label之间的相关性都不是很强。
2.同时,可以发现部分特征存在右偏的现象(skewness (偏度) 描述数据分布形态的统计量,其描述的是某总体取值分布的对称性),考虑到需要数据尽量服从正态分布,所以后续数据预处理需要对存在一定偏度的特征进行相关处理。
# 观察各特征分布和患病的关系 corr = all_data.corr() plt.figure(figsize = (8,6)) sns.heatmap(corr,annot = True,cmap = 'Blues') plt.show()
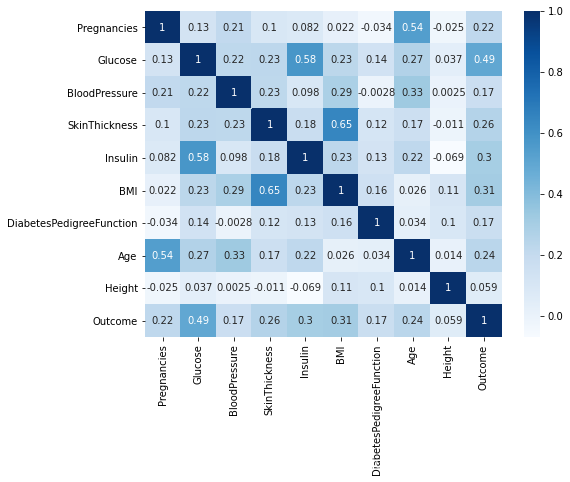
heatmap()函数可以直观地将数据值的大小以定义的颜色深浅表示出来。
1.可以发现颜色相对来说都比较浅,也就是说无论是特征和特征之间还是特征和outcome标签之间的相关性都没有很高。
2.发现其余各特征变量中与outcome的相关度中最高的是Glucose 属性值为0.49,最低的是Height属性值为0.059。
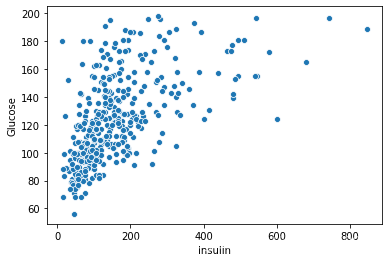
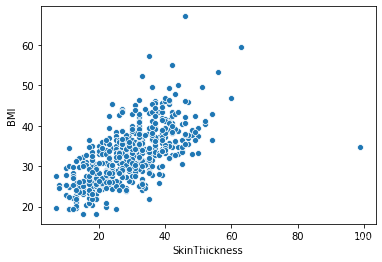
3.同时观察特征与特征之间的关系,发现Insulin与Glucose,BMI和SkinThickness之间的相关度分别为0.58,0.65属于比较高的相关性,由于Insulin是一个确实比较严重的特征,而相关性可以是一种协助填充缺失值的方法。
plt.figure() sns.scatterplot(x = 'Insulin', y = 'Glucose', data = all_data) plt.show() sns.scatterplot(x = 'Insulin', y = 'BMI', data = all_data) plt.show() sns.scatterplot(x = 'Insulin', y = 'Age', data = all_data) plt.show() plt.figure() sns.scatterplot(x = 'SkinThickness', y = 'BMI', data = all_data) plt.show() sns.scatterplot(x = 'SkinThickness', y = 'Glucose', data = all_data) plt.show() sns.scatterplot(x = 'SkinThickness', y = 'BloodPressure', data = all_data) plt.show()
部分输出:
六、构建baseline
因为决策树几乎不需要数据预处理。其他方法经常需要数据标准化,创建虚拟变量和删除缺失值。
# 读取数据
all_data = pd.read_csv('data.csv')
# height 数值类型 为object 需要转化为 数值型
all_data = all_data.astype({'Height':'float64'})
#
all_data.dropna(inplace = True)
# 特征
feature_data = all_data.drop('Outcome',1)
# 标签
label = all_data['Outcome']
base_model = DecisionTreeClassifier()
base_scores = cross_validate(base_model, feature_data, label,cv=5,return_train_score=True)
print(base_scores['test_score'].mean())
0.6954248366013072
七、数据预处理
综合前面分析,先做了以下处理
# 读取数据
all_data = pd.read_csv('data.csv')
# height 数值类型 为object 需要转化为 数值型
all_data = all_data.astype({'Height':'float64'})
# 理论缺失值0替换为np.nan
replace_list = ['Glucose', 'BloodPressure', 'SkinThickness', 'Insulin', 'BMI', 'Height']
all_data.loc[:,replace_list] = all_data.loc[:,replace_list].replace({0:np.nan})
# 删除相关性低的Height
all_data.drop('Height',1,inplace = True)
八、离群值处理
1.经过前面的分析发现数据是存在部分离群值的,虽然实验本身就是关于疾病预测,异常值的存在属于正常现象。但是对于一些可能超出人体接受范围的值,衡量对预测的影响之后,由于数据量比较小,这里选择删除极端异常点。
2.极端异常点 :上限的计算公式为Q3+3(Q3-Q1) 下界的计算公式为Q1-3(Q3-Q1))。
# remove the outliers
# 异常点 上须的计算公式为Q3+1.5(Q3-Q1);下须的计算公式为Q1-1.5(Q3-Q1)
# 极端异常点 :上限的计算公式为Q3+3(Q3-Q1) 下界的计算公式为Q1-3(Q3-Q1)
# 由于数据量比较少 所以选择删除极端异常值
def remove_outliers(feature,all_data):
first_quartile = all_data[feature].describe()['25%']
third_quartile = all_data[feature].describe()['75%']
iqr = third_quartile - first_quartile
# 异常值下标
index = all_data[(all_data[feature] < (first_quartile - 3*iqr)) | (all_data[feature] > (first_quartile + 3*iqr))].index
all_data = all_data.drop(index)
return all_data
outlier_features = ['Insulin', 'Glucose', 'BloodPressure', 'SkinThickness', 'BMI', 'DiabetesPedigreeFunction']
for feat in outlier_features:
all_data = remove_outliers(feat,all_data)
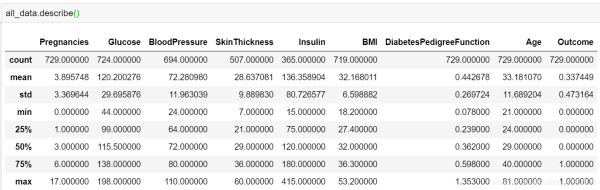
处理之后的数据基本的统计信息
九、缺失值处理
1.直接删除处理
def drop_method(all_data):
median_fill = ['Glucose', 'BloodPressure','SkinThickness', 'BMI','Height']
for column in median_fill:
median_val = all_data[column].median()
all_data[column].fillna(median_val, inplace=True)
all_data.dropna(inplace = True)
return all_data
2.中值填充
def median_method():
for column in list(all_data.columns[all_data.isnull().sum() > 0]):
median = all_data[column].median()
all_data[column].fillna(median, inplace=True)
3.KNNImputer填充
def knn_method():
# 先将缺失值比较少的特征用中值填充
values = {'Glucose': all_data['Glucose'].median(),'BloodPressure':all_data['BloodPressure'].median(),'BMI':all_data['BMI'].median()}
all_data.fillna(value=values,inplace=True)
# 用KNNImputer 填充 Insulin SkinThickness
corr_SkinThickness = ['BMI', 'Glucose','BloodPressure', 'SkinThickness']
# 权重按距离的倒数表示。在这种情况下,查询点的近邻比远处的近邻具有更大的影响力
SkinThickness_imputer = KNNImputer(weights = 'distance')
all_data[corr_SkinThickness] = SkinThickness_imputer.fit_transform(all_data[corr_SkinThickness])
corr_Insulin = ['Glucose', 'BMI','BloodPressure', 'Insulin']
Insulin_imputer = KNNImputer(weights = 'distance')
all_data[corr_Insulin] = Insulin_imputer.fit_transform(all_data[corr_Insulin])
4.随机森林填充
from sklearn.ensemble import RandomForestRegressor
from sklearn.impute import SimpleImputer # 用来填补缺失值
def predict_method(feature):
# 复制一份数据 避免对原数据做出不必要的修改
copy_data = all_data.copy()
# 缺失了的下标
predict_index = copy_data[copy_data[feature].isnull()].index
# 没缺失的下标
train_index = copy_data[feature].dropna().index
# 用作预测 的训练集标签
train_label = copy_data.loc[train_index,feature]
copy_data = copy_data.drop(feature,axis=1)
# 对特征先用中值填充
imp_median = SimpleImputer(strategy='median')
# 用作预测的训练集特征
train_feature = copy_data.loc[train_index]
train_feature = imp_median.fit_transform(train_feature)
# 需要进行预测填充处理的缺失值
pre_feature = copy_data.loc[predict_index]
pre_feature = imp_median.fit_transform(pre_feature)
# 选取随机森林模型
fill_model = RandomForestRegressor()
fill_model = fill_model.fit(train_feature,train_label)
# 预测 填充
pre_value = fill_model.predict(pre_feature)
all_data.loc[predict_index,feature] = pre_value
#用随机森林的方法填充缺失值较多的 SkinThickness 和 Insulin 缺失值
predict_method("Insulin")
predict_method("SkinThickness")
# 其余值中值填充
for column in list(all_data.columns[all_data.isnull().sum() > 0]):
median = all_data[column].median()
all_data[column].fillna(median, inplace=True)
十、特征工程
# 特征
feture_data = all_data.drop('Outcome',1)
# 标签
label = all_data['Outcome']
# 利用BMI和身高构造weight特征 # BMI = weight(kg) / height(m)**2 feture_data['weight'] = (0.01*feture_data['Height'])**2 * feture_data['BMI']
十一、数据标准化
# 标准化 Std = StandardScaler() feture_data = Std.fit_transform(feture_data)
十二、模型构建与调参优化
用到的模型
from sklearn.svm import SVC,SVR
from sklearn.tree import DecisionTreeClassifier
from sklearn.linear_model import LogisticRegression
from sklearn.ensemble import RandomForestClassifier,StackingClassifier
调参方法
from sklearn.model_selection import GridSearchCV
评估指标 Accuracy roc_auc
from sklearn.metrics import make_scorer from sklearn.metrics import
accuracy_score from sklearn.metrics import roc_auc_score
def train(model, params):
grid_search = GridSearchCV(estimator = model, param_grid = params,scoring=scores,refit='Accuracy')
grid_search.fit(feture_data,label)
print(grid_search.best_estimator_)
return grid_search
def paint(x,y):
plt.figure()
sns.lineplot(x=x,y=y)
plt.show()
SVC
#调参时先尝试一个大范围,确定比较小的范围,然后在小范围里搜索
model = SVC()
params = {'C':np.linspace(0.1, 2, 100)}
SVC_grid_search = train(model,params)
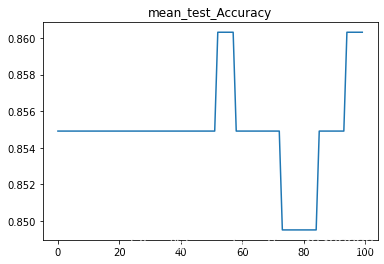
paint([x for x in range(100)],SVC_grid_search.cv_results_['mean_test_Accuracy'])
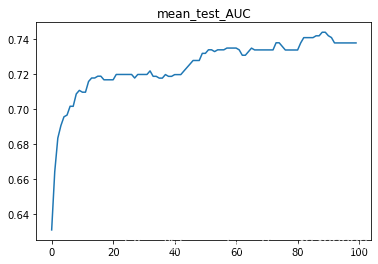
paint([x for x in range(100)],SVC_grid_search.cv_results_['mean_test_AUC'])
print("test_Accuracy : {}\ntest_AUC : {}".format(SVC_grid_search.cv_results_['mean_test_Accuracy'].mean(),SVC_grid_search.cv_results_['mean_test_AUC'].mean()))
LogisticRegression
model = LogisticRegression()
params = {"C":np.linspace(0.1,2,100)}
LR_grid_search = train(model,params)
paint([x for x in range(100)],LR_grid_search.cv_results_['mean_test_Accuracy'])
paint([x for x in range(100)],LR_grid_search.cv_results_['mean_test_AUC'])
print("test_Accuracy : {}\ntest_AUC : {}".format(LR_grid_search.cv_results_['mean_test_Accuracy'].mean(),LR_grid_search.cv_results_['mean_test_AUC'].mean()))
RandomForestClassifier
model = RandomForestClassifier()
params = {"n_estimators":[x for x in range(30,50,2)],'min_samples_split':[x for x in range(4,10)]}
RFC_grid_search = train(model,params)
print("test_Accuracy : {}\ntest_AUC : {}".format(RFC_grid_search.cv_results_['mean_test_Accuracy'].mean(),RFC_grid_search.cv_results_['mean_test_AUC'].mean()))
StackingClassifier
estimators = [
('SVC',SVC_grid_search.best_estimator_),
('NB', LR_grid_search.best_estimator_),
('RFC', RFC_grid_search.best_estimator_)
]
model = StackingClassifier(estimators=estimators, final_estimator=SVC())
model_score = cross_validate(model,feture_data, label,scoring=scores)
print("test_Accuracy : {}\ntest_AUC : {}".format(model_score['test_Accuracy'].mean(),model_score['test_AUC'].mean()))
SVC预测结果:
1.直接删除缺失值以及异常值删除公式上限Q3+1.5(Q3-Q1);下限计算公式为Q1-1.5(Q3-Q1)
SVC(C=1.097979797979798)
test_Accuracy : 0.8549075391180654
test_AUC : 0.511601411290322
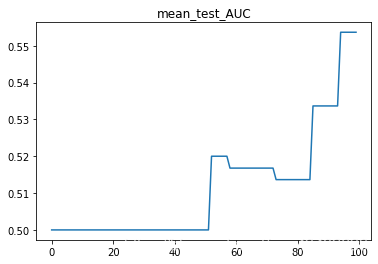
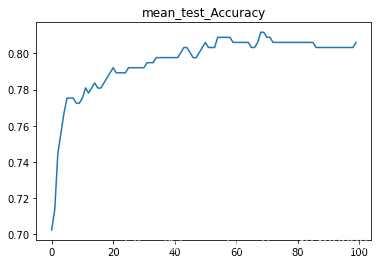
2.直接删除缺失值以及异常值删除公式上限Q3+3(Q3-Q1);下限计算公式为Q1-3(Q3-Q1)
SVC(C=1.405050505050505)
test_Accuracy : 0.7953321596244133
test_AUC : 0.7133755225726653
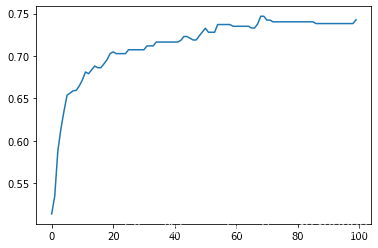
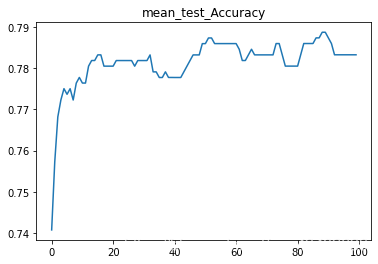
3.中值填充以及异常值删除公式上限Q3+3(Q3-Q1);下限计算公式为Q1-3(Q3-Q1)
SVC(C=1.7888888888888888)
test_Accuracy : 0.7814101086443079
test_AUC : 0.7248522348166069
十三、总结
1.一些删除数据值的处理方法导致样本标签的不均衡会导致对比例大的样本造成过拟合,也就是说预测偏向样本数较多的分类。这样就会大大降低模型的泛化能力。表现在准确率很高,但roc_auc_score很低。上面SVC的预测结果就很好的说明了。
2.可以看出由于缺失值比较多,所以反而各种填充方法的效果比直接删除的效果是要更差的(也有可能我没找到合适的填充方法)
3.关于离群值的处理,主要方法有直接删除法,替换为缺失值处理,以及中值填充法等。由于缺失值处理那里的效果不是很理想,所以就选择了直接删除,并且在平衡了roc_auc_score和accuracy两个指标后,选择只删除极端异常点。
4.关于样本0/1比例的问题,可以考虑上采样或者下采样的方法平衡样本。本文不涉及。
到此这篇关于python数据分析之用sklearn预测糖尿病的文章就介绍到这了,更多相关用sklearn预测糖尿病内容请搜索hwidc以前的文章或继续浏览下面的相关文章希望大家以后多多支持hwidc!
